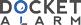`Case 1:20-cv-01644-RGA Document 1-56 Filed 12/03/20 Page 1 of 3 PageID #: 1349
`
`EXHIBIT 56
`
`EXHIBIT 56
`
`
`
`
`
`
`
`Case 1:20-cv-01644-RGA Document 1-56 Filed 12/03/20 Page 2 of 3 PageID #: 1350
`Case 1:20-cv-01644-RGA Document 1-56 Filed 12/03/20 Page 2 of 3_
`gelD #: 1350
`(cid:51)(cid:85)(cid:72)(cid:83)(cid:68)(cid:85)(cid:72) (cid:47)(cid:76)(cid:69)(cid:85)(cid:68)(cid:85)(cid:92)(cid:368)(cid:368)(cid:91)(cid:368)(cid:368)(cid:50)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:68)(cid:368)(cid:368)(cid:91)(cid:368)(cid:368)(cid:32)(cid:77)(cid:64)(cid:75)(cid:88)(cid:89)(cid:68) (cid:35)(cid:64)(cid:83)(cid:64)
`Prepare Library | Sequence | Analyze Data
`I umI na"‘
`
`(cid:55)(cid:85)(cid:88)(cid:54)(cid:76)(cid:74)(cid:75)(cid:87)(cid:289) (cid:50)(cid:81)(cid:70)(cid:82)(cid:79)(cid:82)(cid:74)(cid:92) (cid:56)(cid:48)(cid:44) (cid:53)(cid:72)(cid:68)(cid:74)(cid:72)(cid:81)(cid:87)(cid:86)
`TruSightT'V| Oncology UMI Reagents
`(cid:40)(cid:85)(cid:85)(cid:82)(cid:85) (cid:70)(cid:82)(cid:85)(cid:85)(cid:72)(cid:70)(cid:87)(cid:76)(cid:82)(cid:81) (cid:90)(cid:76)(cid:87)(cid:75) (cid:88)(cid:81)(cid:76)(cid:84)(cid:88)(cid:72) (cid:80)(cid:82)(cid:79)(cid:72)(cid:70)(cid:88)(cid:79)(cid:68)(cid:85) (cid:76)(cid:71)(cid:72)(cid:81)(cid:87)(cid:76)(cid:73)(cid:76)(cid:72)(cid:85)(cid:86) (cid:11)(cid:56)(cid:48)(cid:44)(cid:86)(cid:12) (cid:73)(cid:82)(cid:85) (cid:86)(cid:72)(cid:84)(cid:88)(cid:72)(cid:81)(cid:70)(cid:76)(cid:81)(cid:74) (cid:79)(cid:76)(cid:69)(cid:85)(cid:68)(cid:85)(cid:76)(cid:72)(cid:86)(cid:17)
`Error correction with unique molecular identifiers (UMls) for sequencing libraries.
`
`Without UMI
`
` 933.1
`
`.100
`
`Pasmon
`
`Wltll UMI
`
`."-| ..
`
`, n n:
`PUMIIUII
`
`ctDNA
`
`Figure 1: Background reduction facilitates accurate variant calling —To enable
`(cid:41)(cid:76)(cid:74)(cid:88)(cid:85)(cid:72)(cid:98)(cid:20)(cid:29) (cid:37)(cid:68)(cid:70)(cid:78)(cid:74)(cid:85)(cid:82)(cid:88)(cid:81)(cid:71) (cid:85)(cid:72)(cid:71)(cid:88)(cid:70)(cid:87)(cid:76)(cid:82)(cid:81) (cid:73)(cid:68)(cid:70)(cid:76)(cid:79)(cid:76)(cid:87)(cid:68)(cid:87)(cid:72)(cid:86) (cid:68)(cid:70)(cid:70)(cid:88)(cid:85)(cid:68)(cid:87)(cid:72) (cid:89)(cid:68)(cid:85)(cid:76)(cid:68)(cid:81)(cid:87) (cid:70)(cid:68)(cid:79)(cid:79)(cid:76)(cid:81)(cid:74)(cid:134)(cid:51)(cid:78) (cid:68)(cid:77)(cid:64)(cid:65)(cid:75)(cid:68)
`accurate detection of rare variants, UMls are integrated with error correction
`(cid:64)(cid:66)(cid:66)(cid:84)(cid:81)(cid:64)(cid:83)(cid:68) (cid:67)(cid:68)(cid:83)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:78)(cid:69) (cid:81)(cid:64)(cid:81)(cid:68) (cid:85)(cid:64)(cid:81)(cid:72)(cid:64)(cid:77)(cid:83)(cid:82)(cid:11) (cid:52)(cid:44)(cid:40)(cid:82) (cid:64)(cid:81)(cid:68) (cid:72)(cid:77)(cid:83)(cid:68)(cid:70)(cid:81)(cid:64)(cid:83)(cid:68)(cid:67) (cid:86)(cid:72)(cid:83)(cid:71) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:66)(cid:78)(cid:81)(cid:81)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77)
`soltware enabling true mutations to be distinguished from background noise. The
`(cid:82)(cid:78)(cid:69)(cid:83)(cid:86)(cid:64)(cid:81)(cid:68) (cid:68)(cid:77)(cid:64)(cid:65)(cid:75)(cid:72)(cid:77)(cid:70) (cid:83)(cid:81)(cid:84)(cid:68) (cid:76)(cid:84)(cid:83)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77)(cid:82) (cid:83)(cid:78) (cid:65)(cid:68) (cid:67)(cid:72)(cid:82)(cid:83)(cid:72)(cid:77)(cid:70)(cid:84)(cid:72)(cid:82)(cid:71)(cid:68)(cid:67) (cid:69)(cid:81)(cid:78)(cid:76) (cid:65)(cid:64)(cid:66)(cid:74)(cid:70)(cid:81)(cid:78)(cid:84)(cid:77)(cid:67) (cid:77)(cid:78)(cid:72)(cid:82)(cid:68)(cid:13) (cid:51)(cid:71)(cid:68)
`bottom panel illustrates how, by removing inherent errors that result in false
`(cid:65)(cid:78)(cid:83)(cid:83)(cid:78)(cid:76) (cid:79)(cid:64)(cid:77)(cid:68)(cid:75) (cid:72)(cid:75)(cid:75)(cid:84)(cid:82)(cid:83)(cid:81)(cid:64)(cid:83)(cid:68)(cid:82) (cid:71)(cid:78)(cid:86)(cid:11) (cid:65)(cid:88) (cid:81)(cid:68)(cid:76)(cid:78)(cid:85)(cid:72)(cid:77)(cid:70) (cid:72)(cid:77)(cid:71)(cid:68)(cid:81)(cid:68)(cid:77)(cid:83) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81)(cid:82) (cid:83)(cid:71)(cid:64)(cid:83) (cid:81)(cid:68)(cid:82)(cid:84)(cid:75)(cid:83) (cid:72)(cid:77) (cid:69)(cid:64)(cid:75)(cid:82)(cid:68)
`positives, the reobced error rate allows true mutations (red dots) to be better
`(cid:79)(cid:78)(cid:82)(cid:72)(cid:83)(cid:72)(cid:85)(cid:68)(cid:82)(cid:11) (cid:83)(cid:71)(cid:68) (cid:81)(cid:68)(cid:67)(cid:84)(cid:66)(cid:68)(cid:67) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:81)(cid:64)(cid:83)(cid:68) (cid:64)(cid:75)(cid:75)(cid:78)(cid:86)(cid:82) (cid:83)(cid:81)(cid:84)(cid:68) (cid:76)(cid:84)(cid:83)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77)(cid:82) (cid:7)(cid:81)(cid:68)(cid:67) (cid:67)(cid:78)(cid:83)(cid:82)(cid:8) (cid:83)(cid:78) (cid:65)(cid:68) (cid:65)(cid:68)(cid:83)(cid:83)(cid:68)(cid:81)
`distinguished from background noise (grey dots)
`(cid:67)(cid:72)(cid:82)(cid:83)(cid:72)(cid:77)(cid:70)(cid:84)(cid:72)(cid:82)(cid:71)(cid:68)(cid:67) (cid:69)(cid:81)(cid:78)(cid:76) (cid:65)(cid:64)(cid:66)(cid:74)(cid:70)(cid:81)(cid:78)(cid:84)(cid:77)(cid:67) (cid:77)(cid:78)(cid:72)(cid:82)(cid:68) (cid:7)(cid:70)(cid:81)(cid:68)(cid:88) (cid:67)(cid:78)(cid:83)(cid:82)(cid:8)
`A. Library Preparation
`Y
`
`
`.
`
`‘
`
`End Repair and A-tailing
`
`Highlights
`(cid:43)(cid:76)(cid:74)(cid:75)(cid:79)(cid:76)(cid:74)(cid:75)(cid:87)(cid:86)
`0 Reduced error rate
`(cid:122) (cid:53)(cid:72)(cid:71)(cid:88)(cid:70)(cid:72)(cid:71) (cid:72)(cid:85)(cid:85)(cid:82)(cid:85) (cid:85)(cid:68)(cid:87)(cid:72)
`Enables the reduction of error rates down to 0.007% orlower
`(cid:36)(cid:77)(cid:64)(cid:65)(cid:75)(cid:68)(cid:82) (cid:83)(cid:71)(cid:68) (cid:81)(cid:68)(cid:67)(cid:84)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:78)(cid:69) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:81)(cid:64)(cid:83)(cid:68)(cid:82) (cid:67)(cid:78)(cid:86)(cid:77) (cid:83)(cid:78) (cid:15)(cid:13)(cid:15)(cid:15)(cid:22)(cid:4) (cid:78)(cid:81) (cid:75)(cid:78)(cid:86)(cid:68)(cid:81)
`0
`Integrated with TruSight Oncology DNA library prep and
`(cid:122) (cid:44)(cid:81)(cid:87)(cid:72)(cid:74)(cid:85)(cid:68)(cid:87)(cid:72)(cid:71) (cid:90)(cid:76)(cid:87)(cid:75) (cid:55)(cid:85)(cid:88)(cid:54)(cid:76)(cid:74)(cid:75)(cid:87) (cid:50)(cid:81)(cid:70)(cid:82)(cid:79)(cid:82)(cid:74)(cid:92) (cid:39)(cid:49)(cid:36) (cid:79)(cid:76)(cid:69)(cid:85)(cid:68)(cid:85)(cid:92) (cid:83)(cid:85)(cid:72)(cid:83) (cid:68)(cid:81)(cid:71)
`enrichment assay
`(cid:72)(cid:81)(cid:85)(cid:76)(cid:70)(cid:75)(cid:80)(cid:72)(cid:81)(cid:87) (cid:68)(cid:86)(cid:86)(cid:68)(cid:92)
`Allows creation of UMI-containing sequencing libraries with
`(cid:32)(cid:75)(cid:75)(cid:78)(cid:86)(cid:82) (cid:66)(cid:81)(cid:68)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77) (cid:78)(cid:69) (cid:52)(cid:44)(cid:40)(cid:12)(cid:66)(cid:78)(cid:77)(cid:83)(cid:64)(cid:72)(cid:77)(cid:72)(cid:77)(cid:70) (cid:82)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:72)(cid:77)(cid:70) (cid:75)(cid:72)(cid:65)(cid:81)(cid:64)(cid:81)(cid:72)(cid:68)(cid:82) (cid:86)(cid:72)(cid:83)(cid:71)
`the TruSightTumor170 DNA oligonucleotides (oligos) on cell-
`(cid:83)(cid:71)(cid:68) (cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83) (cid:51)(cid:84)(cid:76)(cid:78)(cid:81) (cid:16)(cid:22)(cid:15) (cid:35)(cid:45)(cid:32) (cid:78)(cid:75)(cid:72)(cid:70)(cid:78)(cid:77)(cid:84)(cid:66)(cid:75)(cid:68)(cid:78)(cid:83)(cid:72)(cid:67)(cid:68)(cid:82) (cid:7)(cid:78)(cid:75)(cid:72)(cid:70)(cid:78)(cid:82)(cid:8)(cid:368)(cid:78)(cid:77) (cid:66)(cid:68)(cid:75)(cid:75)(cid:12)
`free DNA (chNA)
`(cid:69)(cid:81)(cid:68)(cid:68) (cid:35)(cid:45)(cid:32) (cid:7)(cid:66)(cid:69)(cid:35)(cid:45)(cid:32)(cid:8)
`0 Accessible analysis software
`(cid:122) (cid:36)(cid:70)(cid:70)(cid:72)(cid:86)(cid:86)(cid:76)(cid:69)(cid:79)(cid:72) (cid:68)(cid:81)(cid:68)(cid:79)(cid:92)(cid:86)(cid:76)(cid:86) (cid:86)(cid:82)(cid:73)(cid:87)(cid:90)(cid:68)(cid:85)(cid:72)
`Provides error correction software in BaseSpaceTM Sequence
`(cid:47)(cid:81)(cid:78)(cid:85)(cid:72)(cid:67)(cid:68)(cid:82) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:66)(cid:78)(cid:81)(cid:81)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:82)(cid:78)(cid:69)(cid:83)(cid:86)(cid:64)(cid:81)(cid:68) (cid:72)(cid:77) (cid:33)(cid:64)(cid:82)(cid:68)(cid:50)(cid:79)(cid:64)(cid:66)(cid:68)(cid:165) (cid:50)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:68)
`Hub orforlocal installation
`(cid:39)(cid:84)(cid:65) (cid:78)(cid:81) (cid:69)(cid:78)(cid:81) (cid:75)(cid:78)(cid:66)(cid:64)(cid:75) (cid:72)(cid:77)(cid:82)(cid:83)(cid:64)(cid:75)(cid:75)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77)
`
`Introduction
`(cid:44)(cid:81)(cid:87)(cid:85)(cid:82)(cid:71)(cid:88)(cid:70)(cid:87)(cid:76)(cid:82)(cid:81)
`Next-generation sequencing (NGS) has enabled cancer researchers
`(cid:45)(cid:68)(cid:87)(cid:83)(cid:12)(cid:70)(cid:68)(cid:77)(cid:68)(cid:81)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77) (cid:82)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:72)(cid:77)(cid:70) (cid:7)(cid:45)(cid:38)(cid:50)(cid:8) (cid:71)(cid:64)(cid:82) (cid:68)(cid:77)(cid:64)(cid:65)(cid:75)(cid:68)(cid:67) (cid:66)(cid:64)(cid:77)(cid:66)(cid:68)(cid:81) (cid:81)(cid:68)(cid:82)(cid:68)(cid:64)(cid:81)(cid:66)(cid:71)(cid:68)(cid:81)(cid:82)
`to assess numerous genes in a single assay. lllumina technology
`(cid:83)(cid:78) (cid:64)(cid:82)(cid:82)(cid:68)(cid:82)(cid:82) (cid:77)(cid:84)(cid:76)(cid:68)(cid:81)(cid:78)(cid:84)(cid:82) (cid:70)(cid:68)(cid:77)(cid:68)(cid:82) (cid:72)(cid:77) (cid:64) (cid:82)(cid:72)(cid:77)(cid:70)(cid:75)(cid:68) (cid:64)(cid:82)(cid:82)(cid:64)(cid:88)(cid:13) (cid:40)(cid:75)(cid:75)(cid:84)(cid:76)(cid:72)(cid:77)(cid:64) (cid:83)(cid:68)(cid:66)(cid:71)(cid:77)(cid:78)(cid:75)(cid:78)(cid:70)(cid:88)
`provides highly accurate sequencing data, yet any synthesis-based
`(cid:79)(cid:81)(cid:78)(cid:85)(cid:72)(cid:67)(cid:68)(cid:82) (cid:71)(cid:72)(cid:70)(cid:71)(cid:75)(cid:88) (cid:64)(cid:66)(cid:66)(cid:84)(cid:81)(cid:64)(cid:83)(cid:68) (cid:82)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:72)(cid:77)(cid:70) (cid:67)(cid:64)(cid:83)(cid:64)(cid:11) (cid:88)(cid:68)(cid:83) (cid:64)(cid:77)(cid:88) (cid:82)(cid:88)(cid:77)(cid:83)(cid:71)(cid:68)(cid:82)(cid:72)(cid:82)(cid:12)(cid:65)(cid:64)(cid:82)(cid:68)(cid:67)
`method involves inherent errors. Although the error rate is low enough
`(cid:76)(cid:68)(cid:83)(cid:71)(cid:78)(cid:67) (cid:72)(cid:77)(cid:85)(cid:78)(cid:75)(cid:85)(cid:68)(cid:82) (cid:72)(cid:77)(cid:71)(cid:68)(cid:81)(cid:68)(cid:77)(cid:83) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81)(cid:82)(cid:13) (cid:32)(cid:75)(cid:83)(cid:71)(cid:78)(cid:84)(cid:70)(cid:71) (cid:83)(cid:71)(cid:68) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:81)(cid:64)(cid:83)(cid:68) (cid:72)(cid:82) (cid:75)(cid:78)(cid:86) (cid:68)(cid:77)(cid:78)(cid:84)(cid:70)(cid:71)
`(less than 0.5%) to successfully accomplish many NGS-based
`(cid:7)(cid:75)(cid:68)(cid:82)(cid:82) (cid:83)(cid:71)(cid:64)(cid:77) (cid:15)(cid:13)(cid:20)(cid:4)(cid:8) (cid:83)(cid:78) (cid:82)(cid:84)(cid:66)(cid:66)(cid:68)(cid:82)(cid:82)(cid:69)(cid:84)(cid:75)(cid:75)(cid:88) (cid:64)(cid:66)(cid:66)(cid:78)(cid:76)(cid:79)(cid:75)(cid:72)(cid:82)(cid:71) (cid:76)(cid:64)(cid:77)(cid:88) (cid:45)(cid:38)(cid:50)(cid:12)(cid:65)(cid:64)(cid:82)(cid:68)(cid:67)
`applications, new approaches that use noninvasive methods for
`(cid:64)(cid:79)(cid:79)(cid:75)(cid:72)(cid:66)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77)(cid:82)(cid:11) (cid:77)(cid:68)(cid:86) (cid:64)(cid:79)(cid:79)(cid:81)(cid:78)(cid:64)(cid:66)(cid:71)(cid:68)(cid:82) (cid:83)(cid:71)(cid:64)(cid:83) (cid:84)(cid:82)(cid:68) (cid:77)(cid:78)(cid:77)(cid:72)(cid:77)(cid:85)(cid:64)(cid:82)(cid:72)(cid:85)(cid:68) (cid:76)(cid:68)(cid:83)(cid:71)(cid:78)(cid:67)(cid:82) (cid:69)(cid:78)(cid:81)
`sample collection may require a lowererror rate. Forexample,
`(cid:82)(cid:64)(cid:76)(cid:79)(cid:75)(cid:68) (cid:66)(cid:78)(cid:75)(cid:75)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:76)(cid:64)(cid:88) (cid:81)(cid:68)(cid:80)(cid:84)(cid:72)(cid:81)(cid:68) (cid:64) (cid:75)(cid:78)(cid:86)(cid:68)(cid:81) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:81)(cid:64)(cid:83)(cid:68)(cid:13) (cid:37)(cid:78)(cid:81) (cid:68)(cid:87)(cid:64)(cid:76)(cid:79)(cid:75)(cid:68)(cid:11)
`analysis of chNA can be used to detect somatic variants in blood
`(cid:64)(cid:77)(cid:64)(cid:75)(cid:88)(cid:82)(cid:72)(cid:82) (cid:78)(cid:69) (cid:66)(cid:69)(cid:35)(cid:45)(cid:32) (cid:66)(cid:64)(cid:77) (cid:65)(cid:68) (cid:84)(cid:82)(cid:68)(cid:67) (cid:83)(cid:78) (cid:67)(cid:68)(cid:83)(cid:68)(cid:66)(cid:83) (cid:82)(cid:78)(cid:76)(cid:64)(cid:83)(cid:72)(cid:66) (cid:85)(cid:64)(cid:81)(cid:72)(cid:64)(cid:77)(cid:83)(cid:82) (cid:72)(cid:77) (cid:65)(cid:75)(cid:78)(cid:78)(cid:67)
`without the need for biopsy; however, the low percentage of
`(cid:86)(cid:72)(cid:83)(cid:71)(cid:78)(cid:84)(cid:83) (cid:83)(cid:71)(cid:68) (cid:77)(cid:68)(cid:68)(cid:67) (cid:69)(cid:78)(cid:81) (cid:65)(cid:72)(cid:78)(cid:79)(cid:82)(cid:88)(cid:26) (cid:71)(cid:78)(cid:86)(cid:68)(cid:85)(cid:68)(cid:81)(cid:11) (cid:83)(cid:71)(cid:68) (cid:75)(cid:78)(cid:86) (cid:79)(cid:68)(cid:81)(cid:66)(cid:68)(cid:77)(cid:83)(cid:64)(cid:70)(cid:68) (cid:78)(cid:69)
`circulating tumor DNA (ctDNA) within total chNA causes variant
`(cid:66)(cid:72)(cid:81)(cid:66)(cid:84)(cid:75)(cid:64)(cid:83)(cid:72)(cid:77)(cid:70) (cid:83)(cid:84)(cid:76)(cid:78)(cid:81) (cid:35)(cid:45)(cid:32) (cid:7)(cid:66)(cid:83)(cid:35)(cid:45)(cid:32)(cid:8) (cid:86)(cid:72)(cid:83)(cid:71)(cid:72)(cid:77) (cid:83)(cid:78)(cid:83)(cid:64)(cid:75) (cid:66)(cid:69)(cid:35)(cid:45)(cid:32) (cid:66)(cid:64)(cid:84)(cid:82)(cid:68)(cid:82) (cid:85)(cid:64)(cid:81)(cid:72)(cid:64)(cid:77)(cid:83)
`allele frequencies to exist nearthe limit of detection of
`(cid:64)(cid:75)(cid:75)(cid:68)(cid:75)(cid:68) (cid:69)(cid:81)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:72)(cid:68)(cid:82) (cid:83)(cid:78) (cid:68)(cid:87)(cid:72)(cid:82)(cid:83) (cid:77)(cid:68)(cid:64)(cid:81) (cid:83)(cid:71)(cid:68) (cid:75)(cid:72)(cid:76)(cid:72)(cid:83) (cid:78)(cid:69)(cid:368)(cid:67)(cid:68)(cid:83)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:78)(cid:69)
`existing methods.
`(cid:68)(cid:87)(cid:72)(cid:82)(cid:83)(cid:72)(cid:77)(cid:70)(cid:368)(cid:76)(cid:68)(cid:83)(cid:71)(cid:78)(cid:67)(cid:82)(cid:13)
`
`To address this challenge, lllumina offers the TruSight Oncology UMI
`(cid:51)(cid:78) (cid:64)(cid:67)(cid:67)(cid:81)(cid:68)(cid:82)(cid:82) (cid:83)(cid:71)(cid:72)(cid:82) (cid:66)(cid:71)(cid:64)(cid:75)(cid:75)(cid:68)(cid:77)(cid:70)(cid:68)(cid:11) (cid:40)(cid:75)(cid:75)(cid:84)(cid:76)(cid:72)(cid:77)(cid:64) (cid:78)(cid:69)(cid:69)(cid:68)(cid:81)(cid:82) (cid:83)(cid:71)(cid:68) (cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83) (cid:46)(cid:77)(cid:66)(cid:78)(cid:75)(cid:78)(cid:70)(cid:88) (cid:52)(cid:44)(cid:40)
`Reagents, which implement UMls and errorcorrection software to
`(cid:49)(cid:68)(cid:64)(cid:70)(cid:68)(cid:77)(cid:83)(cid:82)(cid:11) (cid:86)(cid:71)(cid:72)(cid:66)(cid:71) (cid:72)(cid:76)(cid:79)(cid:75)(cid:68)(cid:76)(cid:68)(cid:77)(cid:83) (cid:52)(cid:44)(cid:40)(cid:82) (cid:64)(cid:77)(cid:67) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:66)(cid:78)(cid:81)(cid:81)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:82)(cid:78)(cid:69)(cid:83)(cid:86)(cid:64)(cid:81)(cid:68) (cid:83)(cid:78)
`lowerthe rate of inherenterrors in NGS data. The UMI reagents
`(cid:75)(cid:78)(cid:86)(cid:68)(cid:81) (cid:83)(cid:71)(cid:68) (cid:81)(cid:64)(cid:83)(cid:68) (cid:78)(cid:69) (cid:72)(cid:77)(cid:71)(cid:68)(cid:81)(cid:68)(cid:77)(cid:83) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81)(cid:82) (cid:72)(cid:77) (cid:45)(cid:38)(cid:50)(cid:368)(cid:67)(cid:64)(cid:83)(cid:64)(cid:13) (cid:51)(cid:71)(cid:68) (cid:52)(cid:44)(cid:40) (cid:81)(cid:68)(cid:64)(cid:70)(cid:68)(cid:77)(cid:83)(cid:82)
`integrate easily with the TruSight Oncology workflow1 by simply
`(cid:72)(cid:77)(cid:83)(cid:68)(cid:70)(cid:81)(cid:64)(cid:83)(cid:68) (cid:68)(cid:64)(cid:82)(cid:72)(cid:75)(cid:88) (cid:86)(cid:72)(cid:83)(cid:71) (cid:83)(cid:71)(cid:68) (cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83) (cid:46)(cid:77)(cid:66)(cid:78)(cid:75)(cid:78)(cid:70)(cid:88) (cid:86)(cid:78)(cid:81)(cid:74)(cid:69)(cid:75)(cid:78)(cid:86)(cid:16) (cid:65)(cid:88) (cid:82)(cid:72)(cid:76)(cid:79)(cid:75)(cid:88)
`replacing standard Y-shaped adapters with UMI-containing adapters
`(cid:81)(cid:68)(cid:79)(cid:75)(cid:64)(cid:66)(cid:72)(cid:77)(cid:70) (cid:82)(cid:83)(cid:64)(cid:77)(cid:67)(cid:64)(cid:81)(cid:67) (cid:56)(cid:12)(cid:82)(cid:71)(cid:64)(cid:79)(cid:68)(cid:67) (cid:64)(cid:67)(cid:64)(cid:79)(cid:83)(cid:68)(cid:81)(cid:82) (cid:86)(cid:72)(cid:83)(cid:71) (cid:52)(cid:44)(cid:40)(cid:12)(cid:66)(cid:78)(cid:77)(cid:83)(cid:64)(cid:72)(cid:77)(cid:72)(cid:77)(cid:70) (cid:64)(cid:67)(cid:64)(cid:79)(cid:83)(cid:68)(cid:81)(cid:82)
`to barcode each individual DNA strand. Reducing the background
`(cid:83)(cid:78) (cid:65)(cid:64)(cid:81)(cid:66)(cid:78)(cid:67)(cid:68) (cid:68)(cid:64)(cid:66)(cid:71) (cid:72)(cid:77)(cid:67)(cid:72)(cid:85)(cid:72)(cid:67)(cid:84)(cid:64)(cid:75) (cid:35)(cid:45)(cid:32) (cid:82)(cid:83)(cid:81)(cid:64)(cid:77)(cid:67)(cid:13) (cid:49)(cid:68)(cid:67)(cid:84)(cid:66)(cid:72)(cid:77)(cid:70) (cid:83)(cid:71)(cid:68) (cid:65)(cid:64)(cid:66)(cid:74)(cid:70)(cid:81)(cid:78)(cid:84)(cid:77)(cid:67)
`noise leadsto a lowerlimit of detection (Figure 1). With the TruSight
`(cid:77)(cid:78)(cid:72)(cid:82)(cid:68) (cid:75)(cid:68)(cid:64)(cid:67)(cid:82) (cid:83)(cid:78) (cid:64) (cid:75)(cid:78)(cid:86)(cid:68)(cid:81) (cid:75)(cid:72)(cid:76)(cid:72)(cid:83) (cid:78)(cid:69) (cid:67)(cid:68)(cid:83)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:7)(cid:37)(cid:72)(cid:70)(cid:84)(cid:81)(cid:68) (cid:16)(cid:8)(cid:13) (cid:54)(cid:72)(cid:83)(cid:71) (cid:83)(cid:71)(cid:68) (cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83)
`Tumor 1 70 DNA assay,2 the TruSight Oncology UMI Reagents
`(cid:51)(cid:84)(cid:76)(cid:78)(cid:81) (cid:16)(cid:22)(cid:15) (cid:35)(cid:45)(cid:32) (cid:64)(cid:82)(cid:82)(cid:64)(cid:88)(cid:11)(cid:17) (cid:83)(cid:71)(cid:68) (cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83) (cid:46)(cid:77)(cid:66)(cid:78)(cid:75)(cid:78)(cid:70)(cid:88) (cid:52)(cid:44)(cid:40) (cid:49)(cid:68)(cid:64)(cid:70)(cid:68)(cid:77)(cid:83)(cid:82)
`reduced the error rate to 5 0.007% or lower. Lowererror rates
`(cid:81)(cid:68)(cid:67)(cid:84)(cid:66)(cid:68)(cid:67) (cid:83)(cid:71)(cid:68) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:81)(cid:64)(cid:83)(cid:68) (cid:83)(cid:78) (cid:173) (cid:15)(cid:13)(cid:15)(cid:15)(cid:22)(cid:4) (cid:78)(cid:81) (cid:75)(cid:78)(cid:86)(cid:68)(cid:81)(cid:13) (cid:43)(cid:78)(cid:86)(cid:68)(cid:81) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:81)(cid:64)(cid:83)(cid:68)(cid:82)
`increase analytical specificity, and the resulting higherconfidence in
`(cid:72)(cid:77)(cid:66)(cid:81)(cid:68)(cid:64)(cid:82)(cid:68) (cid:64)(cid:77)(cid:64)(cid:75)(cid:88)(cid:83)(cid:72)(cid:66)(cid:64)(cid:75) (cid:82)(cid:79)(cid:68)(cid:66)(cid:72)(cid:69)(cid:72)(cid:66)(cid:72)(cid:83)(cid:88)(cid:11) (cid:64)(cid:77)(cid:67) (cid:83)(cid:71)(cid:68) (cid:81)(cid:68)(cid:82)(cid:84)(cid:75)(cid:83)(cid:72)(cid:77)(cid:70) (cid:71)(cid:72)(cid:70)(cid:71)(cid:68)(cid:81) (cid:66)(cid:78)(cid:77)(cid:69)(cid:72)(cid:67)(cid:68)(cid:77)(cid:66)(cid:68) (cid:72)(cid:77)
`variant calls help put ctDNA analysis into the hands of translational
`(cid:85)(cid:64)(cid:81)(cid:72)(cid:64)(cid:77)(cid:83) (cid:66)(cid:64)(cid:75)(cid:75)(cid:82) (cid:71)(cid:68)(cid:75)(cid:79) (cid:79)(cid:84)(cid:83) (cid:66)(cid:83)(cid:35)(cid:45)(cid:32) (cid:64)(cid:77)(cid:64)(cid:75)(cid:88)(cid:82)(cid:72)(cid:82) (cid:72)(cid:77)(cid:83)(cid:78) (cid:83)(cid:71)(cid:68) (cid:71)(cid:64)(cid:77)(cid:67)(cid:82) (cid:78)(cid:69) (cid:83)(cid:81)(cid:64)(cid:77)(cid:82)(cid:75)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77)(cid:64)(cid:75)
`cancer researchers.
`(cid:66)(cid:64)(cid:77)(cid:66)(cid:68)(cid:81)(cid:368)(cid:81)(cid:68)(cid:82)(cid:68)(cid:64)(cid:81)(cid:66)(cid:71)(cid:68)(cid:81)(cid:82)(cid:13)
`
`(cid:54)(cid:76)(cid:80)(cid:83)(cid:79)(cid:72) (cid:76)(cid:81)(cid:87)(cid:72)(cid:74)(cid:85)(cid:68)(cid:87)(cid:76)(cid:82)(cid:81) (cid:76)(cid:81)(cid:87)(cid:82) (cid:87)(cid:75)(cid:72) (cid:55)(cid:85)(cid:88)(cid:54)(cid:76)(cid:74)(cid:75)(cid:87)
`Simple integration into the TruSight
`(cid:50)(cid:81)(cid:70)(cid:82)(cid:79)(cid:82)(cid:74)(cid:92)(cid:98)(cid:90)(cid:82)(cid:85)(cid:78)(cid:73)(cid:79)(cid:82)(cid:90)
`Oncology workflow
`The TruSight Oncology UMI Reagents provide UMI-adapters and
`(cid:51)(cid:71)(cid:68) (cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83) (cid:46)(cid:77)(cid:66)(cid:78)(cid:75)(cid:78)(cid:70)(cid:88) (cid:52)(cid:44)(cid:40) (cid:49)(cid:68)(cid:64)(cid:70)(cid:68)(cid:77)(cid:83)(cid:82) (cid:79)(cid:81)(cid:78)(cid:85)(cid:72)(cid:67)(cid:68) (cid:52)(cid:44)(cid:40)(cid:12)(cid:64)(cid:67)(cid:64)(cid:79)(cid:83)(cid:68)(cid:81)(cid:82) (cid:64)(cid:77)(cid:67)
`indexes, plus TruSight Oncology DNA library prep and enrichment
`(cid:72)(cid:77)(cid:67)(cid:68)(cid:87)(cid:68)(cid:82)(cid:11) (cid:79)(cid:75)(cid:84)(cid:82) (cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83) (cid:46)(cid:77)(cid:66)(cid:78)(cid:75)(cid:78)(cid:70)(cid:88) (cid:35)(cid:45)(cid:32) (cid:75)(cid:72)(cid:65)(cid:81)(cid:64)(cid:81)(cid:88) (cid:79)(cid:81)(cid:68)(cid:79) (cid:64)(cid:77)(cid:67) (cid:68)(cid:77)(cid:81)(cid:72)(cid:66)(cid:71)(cid:76)(cid:68)(cid:77)(cid:83)
`reagents, that can be paired with the TruSight Tumor 1 70 DNA oligos
`(cid:81)(cid:68)(cid:64)(cid:70)(cid:68)(cid:77)(cid:83)(cid:82)(cid:11) (cid:83)(cid:71)(cid:64)(cid:83) (cid:66)(cid:64)(cid:77) (cid:65)(cid:68) (cid:79)(cid:64)(cid:72)(cid:81)(cid:68)(cid:67) (cid:86)(cid:72)(cid:83)(cid:71) (cid:83)(cid:71)(cid:68) (cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83) (cid:51)(cid:84)(cid:76)(cid:78)(cid:81) (cid:16)(cid:22)(cid:15) (cid:35)(cid:45)(cid:32) (cid:78)(cid:75)(cid:72)(cid:70)(cid:78)(cid:82)
`to create UMI-containing sequencing libraries. By replacing the
`(cid:83)(cid:78) (cid:66)(cid:81)(cid:68)(cid:64)(cid:83)(cid:68) (cid:52)(cid:44)(cid:40)(cid:12)(cid:66)(cid:78)(cid:77)(cid:83)(cid:64)(cid:72)(cid:77)(cid:72)(cid:77)(cid:70) (cid:82)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:72)(cid:77)(cid:70) (cid:75)(cid:72)(cid:65)(cid:81)(cid:64)(cid:81)(cid:72)(cid:68)(cid:82)(cid:13) (cid:33)(cid:88) (cid:81)(cid:68)(cid:79)(cid:75)(cid:64)(cid:66)(cid:72)(cid:77)(cid:70) (cid:83)(cid:71)(cid:68)
`standard adapters with the UMI-containing adapters during the
`(cid:82)(cid:83)(cid:64)(cid:77)(cid:67)(cid:64)(cid:81)(cid:67) (cid:64)(cid:67)(cid:64)(cid:79)(cid:83)(cid:68)(cid:81)(cid:82) (cid:86)(cid:72)(cid:83)(cid:71) (cid:83)(cid:71)(cid:68) (cid:52)(cid:44)(cid:40)(cid:12)(cid:66)(cid:78)(cid:77)(cid:83)(cid:64)(cid:72)(cid:77)(cid:72)(cid:77)(cid:70) (cid:64)(cid:67)(cid:64)(cid:79)(cid:83)(cid:68)(cid:81)(cid:82) (cid:67)(cid:84)(cid:81)(cid:72)(cid:77)(cid:70) (cid:83)(cid:71)(cid:68)
`ligation step, integration of UMls does not create any extra steps in the
`(cid:75)(cid:72)(cid:70)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77) (cid:82)(cid:83)(cid:68)(cid:79)(cid:11) (cid:72)(cid:77)(cid:83)(cid:68)(cid:70)(cid:81)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77) (cid:78)(cid:69) (cid:52)(cid:44)(cid:40)(cid:82) (cid:67)(cid:78)(cid:68)(cid:82) (cid:77)(cid:78)(cid:83) (cid:66)(cid:81)(cid:68)(cid:64)(cid:83)(cid:68) (cid:64)(cid:77)(cid:88) (cid:68)(cid:87)(cid:83)(cid:81)(cid:64) (cid:82)(cid:83)(cid:68)(cid:79)(cid:82) (cid:72)(cid:77) (cid:83)(cid:71)(cid:68)
`library prep workflow (Figure 2).
`(cid:75)(cid:72)(cid:65)(cid:81)(cid:64)(cid:81)(cid:88) (cid:79)(cid:81)(cid:68)(cid:79) (cid:86)(cid:78)(cid:81)(cid:74)(cid:69)(cid:75)(cid:78)(cid:86) (cid:7)(cid:37)(cid:72)(cid:70)(cid:84)(cid:81)(cid:68) (cid:17)(cid:8)(cid:13)
`
`Y
`
`
`
`Y
`
`n—unu
`_""'
`
`a
`
`==
`
`A
`
`um _ UMI-containing
`"1"" _ adapter ligation
`f IrIU'Jx
`finl = Index PER
`
`Illllfix I
`_ mm .— uu
`— Inco- _ um
`
`v
`
`7
`
`our _ w.» _ UMI-containing
`um _. «an. _ library
`
`B. Target Enrichment
`T
`
`C. Sequencing
`Y
`
`D. Analysis with UMI—based Error Correction Software
`
`Figure 2: Target capture library prep wo rkflow— UMls can be integrated into
`(cid:41)(cid:76)(cid:74)(cid:88)(cid:85)(cid:72)(cid:98)(cid:21)(cid:29) (cid:55)(cid:68)(cid:85)(cid:74)(cid:72)(cid:87) (cid:70)(cid:68)(cid:83)(cid:87)(cid:88)(cid:85)(cid:72) (cid:79)(cid:76)(cid:69)(cid:85)(cid:68)(cid:85)(cid:92) (cid:83)(cid:85)(cid:72)(cid:83) (cid:90)(cid:82)(cid:85)(cid:78)(cid:73)(cid:79)(cid:82)(cid:90)(cid:134)(cid:52)(cid:44)(cid:40)(cid:82) (cid:66)(cid:64)(cid:77) (cid:65)(cid:68) (cid:72)(cid:77)(cid:83)(cid:68)(cid:70)(cid:81)(cid:64)(cid:83)(cid:68)(cid:67) (cid:72)(cid:77)(cid:83)(cid:78)
`TruSight Oncolong—adapter ligation without interrupting the standard workflow.
`(cid:51)(cid:81)(cid:84)(cid:50)(cid:72)(cid:70)(cid:71)(cid:83) (cid:46)(cid:77)(cid:66)(cid:78)(cid:75)(cid:78)(cid:70)(cid:88) (cid:56)(cid:12)(cid:64)(cid:67)(cid:64)(cid:79)(cid:83)(cid:68)(cid:81) (cid:75)(cid:72)(cid:70)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77) (cid:86)(cid:72)(cid:83)(cid:71)(cid:78)(cid:84)(cid:83) (cid:72)(cid:77)(cid:83)(cid:68)(cid:81)(cid:81)(cid:84)(cid:79)(cid:83)(cid:72)(cid:77)(cid:70) (cid:83)(cid:71)(cid:68) (cid:82)(cid:83)(cid:64)(cid:77)(cid:67)(cid:64)(cid:81)(cid:67) (cid:86)(cid:78)(cid:81)(cid:74)(cid:69)(cid:75)(cid:78)(cid:86)(cid:13)
`During library preparation, the aobpters from the original library prep kit are simply
`(cid:35)(cid:84)(cid:81)(cid:72)(cid:77)(cid:70) (cid:75)(cid:72)(cid:65)(cid:81)(cid:64)(cid:81)(cid:88) (cid:79)(cid:81)(cid:68)(cid:79)(cid:64)(cid:81)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77)(cid:11) (cid:83)(cid:71)(cid:68) (cid:64)(cid:67)(cid:64)(cid:79)(cid:83)(cid:68)(cid:81)(cid:82) (cid:69)(cid:81)(cid:78)(cid:76) (cid:83)(cid:71)(cid:68) (cid:78)(cid:81)(cid:72)(cid:70)(cid:72)(cid:77)(cid:64)(cid:75) (cid:75)(cid:72)(cid:65)(cid:81)(cid:64)(cid:81)(cid:88) (cid:79)(cid:81)(cid:68)(cid:79) (cid:74)(cid:72)(cid:83) (cid:64)(cid:81)(cid:68) (cid:82)(cid:72)(cid:76)(cid:79)(cid:75)(cid:88)
`replaced by the UMI aobpters
`(cid:81)(cid:68)(cid:79)(cid:75)(cid:64)(cid:66)(cid:68)(cid:67) (cid:65)(cid:88) (cid:83)(cid:71)(cid:68) (cid:52)(cid:44)(cid:40) (cid:64)(cid:67)(cid:64)(cid:79)(cid:83)(cid:68)(cid:81)(cid:82)(cid:13)
`
`For Research Use Only. Not for use in diagnostic procedures.
`(cid:41)(cid:82)(cid:85) (cid:53)(cid:72)(cid:86)(cid:72)(cid:68)(cid:85)(cid:70)(cid:75) (cid:56)(cid:86)(cid:72) (cid:50)(cid:81)(cid:79)(cid:92)(cid:17) (cid:49)(cid:82)(cid:87) (cid:73)(cid:82)(cid:85) (cid:88)(cid:86)(cid:72) (cid:76)(cid:81) (cid:71)(cid:76)(cid:68)(cid:74)(cid:81)(cid:82)(cid:86)(cid:87)(cid:76)(cid:70) (cid:83)(cid:85)(cid:82)(cid:70)(cid:72)(cid:71)(cid:88)(cid:85)(cid:72)(cid:86)(cid:17)
`
`1000000050425 VOO |
`1
`(cid:16)(cid:15)(cid:15)(cid:15)(cid:15)(cid:15)(cid:15)(cid:15)(cid:20)(cid:15)(cid:19)(cid:17)(cid:20) (cid:85)(cid:15)(cid:15) (cid:368)(cid:91)(cid:368)(cid:368)(cid:16)
`
`
`
`Case 1:20-cv-01644-RGA Document 1-56 Filed 12/03/20 Page 3 of 3 PageID #: 1351
`Case 1:20-cv-01644-RGA Document 1-56 Filed 12/03/20 Page 3 of 3 PagelD #: 1351
`(cid:51)(cid:85)(cid:72)(cid:83)(cid:68)(cid:85)(cid:72) (cid:47)(cid:76)(cid:69)(cid:85)(cid:68)(cid:85)(cid:92)(cid:368)(cid:368)(cid:91)(cid:368)(cid:368)(cid:50)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:68)(cid:368)(cid:368)(cid:91)(cid:368)(cid:368)(cid:32)(cid:77)(cid:64)(cid:75)(cid:88)(cid:89)(cid:68) (cid:35)(cid:64)(cid:83)(cid:64)
`Prepare Library | Sequence | Analyze Data
`
`Intuitive software for error correction
`(cid:44)(cid:81)(cid:87)(cid:88)(cid:76)(cid:87)(cid:76)(cid:89)(cid:72) (cid:86)(cid:82)(cid:73)(cid:87)(cid:90)(cid:68)(cid:85)(cid:72) (cid:73)(cid:82)(cid:85) (cid:72)(cid:85)(cid:85)(cid:82)(cid:85) (cid:70)(cid:82)(cid:85)(cid:85)(cid:72)(cid:70)(cid:87)(cid:76)(cid:82)(cid:81)
`After UMI-containing libraries are sequenced, the UMI Error
`(cid:32)(cid:69)(cid:83)(cid:68)(cid:81) (cid:52)(cid:44)(cid:40)(cid:12)(cid:66)(cid:78)(cid:77)(cid:83)(cid:64)(cid:72)(cid:77)(cid:72)(cid:77)(cid:70) (cid:75)(cid:72)(cid:65)(cid:81)(cid:64)(cid:81)(cid:72)(cid:68)(cid:82) (cid:64)(cid:81)(cid:68) (cid:82)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:68)(cid:67)(cid:11) (cid:83)(cid:71)(cid:68) (cid:52)(cid:44)(cid:40) (cid:36)(cid:81)(cid:81)(cid:78)(cid:81)
`Correction App aligns reads, then collapses the sequences with
`(cid:34)(cid:78)(cid:81)(cid:81)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:32)(cid:79)(cid:79) (cid:64)(cid:75)(cid:72)(cid:70)(cid:77)(cid:82) (cid:81)(cid:68)(cid:64)(cid:67)(cid:82)(cid:11) (cid:83)(cid:71)(cid:68)(cid:77) (cid:66)(cid:78)(cid:75)(cid:75)(cid:64)(cid:79)(cid:82)(cid:68)(cid:82) (cid:83)(cid:71)(cid:68) (cid:82)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:68)(cid:82) (cid:86)(cid:72)(cid:83)(cid:71)
`shared UMls down to unique reads (Figure 3). This process enables
`(cid:82)(cid:71)(cid:64)(cid:81)(cid:68)(cid:67) (cid:52)(cid:44)(cid:40)(cid:82) (cid:67)(cid:78)(cid:86)(cid:77) (cid:83)(cid:78) (cid:84)(cid:77)(cid:72)(cid:80)(cid:84)(cid:68) (cid:81)(cid:68)(cid:64)(cid:67)(cid:82) (cid:7)(cid:37)(cid:72)(cid:70)(cid:84)(cid:81)(cid:68) (cid:18)(cid:8)(cid:13) (cid:51)(cid:71)(cid:72)(cid:82) (cid:79)(cid:81)(cid:78)(cid:66)(cid:68)(cid:82)(cid:82) (cid:68)(cid:77)(cid:64)(cid:65)(cid:75)(cid:68)(cid:82)
`the filtering of false positives, which are excluded from the collapsed
`(cid:83)(cid:71)(cid:68) (cid:69)(cid:72)(cid:75)(cid:83)(cid:68)(cid:81)(cid:72)(cid:77)(cid:70) (cid:78)(cid:69) (cid:69)(cid:64)(cid:75)(cid:82)(cid:68) (cid:79)(cid:78)(cid:82)(cid:72)(cid:83)(cid:72)(cid:85)(cid:68)(cid:82)(cid:11) (cid:86)(cid:71)(cid:72)(cid:66)(cid:71) (cid:64)(cid:81)(cid:68) (cid:68)(cid:87)(cid:66)(cid:75)(cid:84)(cid:67)(cid:68)(cid:67) (cid:69)(cid:81)(cid:78)(cid:76) (cid:83)(cid:71)(cid:68) (cid:66)(cid:78)(cid:75)(cid:75)(cid:64)(cid:79)(cid:82)(cid:68)(cid:67)
`reads, reducing the error rate for variant calling (Table 1). The UMI
`(cid:81)(cid:68)(cid:64)(cid:67)(cid:82)(cid:11) (cid:81)(cid:68)(cid:67)(cid:84)(cid:66)(cid:72)(cid:77)(cid:70) (cid:83)(cid:71)(cid:68) (cid:68)(cid:81)(cid:81)(cid:78)(cid:81) (cid:81)(cid:64)(cid:83)(cid:68) (cid:69)(cid:78)(cid:81) (cid:85)(cid:64)(cid:81)(cid:72)(cid:64)(cid:77)(cid:83) (cid:66)(cid:64)(cid:75)(cid:75)(cid:72)(cid:77)(cid:70) (cid:7)(cid:51)(cid:64)(cid:65)(cid:75)(cid:68) (cid:16)(cid:8)(cid:13) (cid:51)(cid:71)(cid:68) (cid:52)(cid:44)(cid:40)
`Error Correction App is available in the cloud-based BaseSpace
`(cid:36)(cid:81)(cid:81)(cid:78)(cid:81) (cid:34)(cid:78)(cid:81)(cid:81)(cid:68)(cid:66)(cid:83)(cid:72)(cid:78)(cid:77) (cid:32)(cid:79)(cid:79) (cid:72)(cid:82) (cid:64)(cid:85)(cid:64)(cid:72)(cid:75)(cid:64)(cid:65)(cid:75)(cid:68) (cid:72)(cid:77) (cid:83)(cid:71)(cid:68) (cid:66)(cid:75)(cid:78)(cid:84)(cid:67)(cid:12)(cid:65)(cid:64)(cid:82)(cid:68)(cid:67) (cid:33)(cid:64)(cid:82)(cid:68)(cid:50)(cid:79)(cid:64)(cid:66)(cid:68)
`Sequence Hub, or for installation on a local workstation.
`(cid:50)(cid:68)(cid:80)(cid:84)(cid:68)(cid:77)(cid:66)(cid:68) (cid:39)(cid:84)(cid:65)(cid:11) (cid:78)(cid:81) (cid:69)(cid:78)(cid:81) (cid:72)(cid:77)(cid:82)(cid:83)(cid:64)(cid:75)(cid:75)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77) (cid:78)(cid:77) (cid:64) (cid:75)(cid:78)(cid:66)(cid:64)(cid:75) (cid:86)(cid:78)(cid:81)(cid:74)(cid:82)(cid:83)(cid:64)(cid:83)(cid:72)(cid:78)(cid:77)(cid:13)
`
`Table 1: Reduction of error rates with UMls
`(cid:55)(cid:68)(cid:69)(cid:79)(cid:72)(cid:98)(cid:20)(cid:29) (cid:53)(cid:72)(cid:71)(cid:88)(cid:70)(cid:87)(cid:76)(cid:82)(cid:81) (cid:82)(cid:73) (cid:72)(cid:85)(cid:85)(cid:82)(cid:85) (cid:85)(cid:68)(cid:87)(cid:72)(cid:86) (cid:90)(cid:76)(cid:87)(cid:75) (cid:56)(cid:48)(cid:44)(cid:86)
`Mean Error Rate
`Sequencing
`Mean Error Rate
`(cid:48)(cid:72)(cid:68)(cid:81) (cid:40)(cid:85)(cid:85)(cid:82)(cid:85) (cid:53)(cid:68)(cid:87)(cid:72)
`(cid:48)(cid:72)(cid:68)(cid:81) (cid:40)(cid:85)(cid:85)(cid:82)(cid:85) (cid:53)(cid:68)(cid:87)(cid:72)
`(cid:54)(cid:72)(cid:84)(cid:88)(cid:72)(cid:81)(cid:70)(cid:76)(cid:81)(cid:74)
`(Collapsed Reads)
`Run
`